近日,由中國科學院海洋研究所張國范研究組獨立完成的論文“Construction of a chromosome-level genome and variation map for the Pacific oyster Crassostrea gigas(長牡蠣染色體水平基因組和變異圖譜構建)”在國際學術期刊Molecular Ecology Resources(《分子生態資源》)在線發表。該研究首次構建了長牡蠣染色體水平基因組和貝類中第一張包含SNP、INDEL和CNV的綜合序列變異圖譜,發現長牡蠣基因組中存在高比例的長片段重復序列,CNV結構變異可導致個體間平均21%的區域差異,更新了長牡蠣基因組資源和對基因組復雜性的認知。
長牡蠣基因組序列圖譜
長牡蠣(Crassostrea gigas),俗稱太平洋牡蠣,是世界重要的養殖貝類,具有重要的經濟和生態價值。張國范研究組長期從事貝類養殖和育種相關工作,并一直致力于牡蠣基因資源挖掘和分子育種研究。2012年在Nature發表的牡蠣第一張基因組圖譜得到廣泛應用,引領并開啟了水產物種基因組學相關研究。張國范研究組之后一直對牡蠣基因資源進行維護和更新。隨著新一代測序技術的普及和對長牡蠣染色體水平基因組需求的增加,研究組于2019年正式啟動長牡蠣染色體水平基因組構建工作。

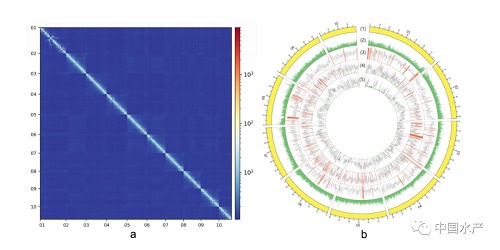
長牡蠣基因組變異圖譜
本研究利用高覆蓋第三代測序(PacBio,~200×)結合HIC技術,并利用遺傳連鎖圖譜進行輔助,對一個中國青島的長牡蠣進行從頭組裝,獲得基因組總長度為587M,包含10條染色體水平scaffold序列,ContigN50達到3.1M,ScaffoldN50達到60M,組裝完整性和連續性均有較大提高。研究發現基因組中高比例的長片段重復在基因進化中發揮了重要作用,同時也是導致之前基于二代測序技術組裝的長牡蠣基因組碎片化的主要原因。研究中基于495個野生長牡蠣的重測序數據分析,構建了長牡蠣綜合變異圖譜,包含480萬個高質量SNP,60萬個INDEL和4.9萬個CNV。這些新的數據資源對牡蠣比較基因組學、分子育種和適應進化等研究具有重要意義。美國南加州大學Dennis Hedgecock教授團隊成員Xiaoshen Yin博士對該基因組進行了分析,發現和連鎖圖譜一致性達到90%以上,明顯的組裝錯誤不到0.1%,肯定了其較高的組裝質量(Yin et al, 2020)。
本研究得到國家自然科學基金、山東省科技創新重大專項、中科院海洋大科學研究中心重點部署項目、國家貝類產業技術體系專項基金等資助。中科院海洋所高性能計算中心對部分生物信息分析提供了技術支持。亓海剛副研究員為論文第一作者,李莉研究員和張國范研究員為通訊作者。
論文鏈接:onlinelibrary.wiley.com/doi/abs/10.1111/1755-0998.13368
| 發表評論 |
咨詢:0779-2029779
隨時,隨地,伴你身邊!
圖文推薦
最新綜合新聞
今日要聞
熱點推薦
X
